1. Bevezetés¶
1.1 Telepítés¶
A csomag sikeres telepítéséhez legalább 3.6.x verziójú Python szükséges.
Szükséges csomagok:
numpy
scipy
pandas
matplotlib
Jinja2
Opcionális csomagok:
lmfit - a részletes illesztési statisztikák miatt
numba - a non-uniform FFT gyorsításához
pytest - a tesztek futtatásáért
Dask - a WFT módszer parallel futtatásáért
scikit-learn - a RANSAC filter miatt
1.1.1 Anaconda környezet¶
Anaconda esetén a telepítés az Anaconda Prompt megnyitásával zajlik. Ahhoz, hogy az Anaconda ne kérdezzen rá minden alkalommal, hogy biztosan telepíteni szeretnénk-e, célszerű futtatni a
conda config --set always_yes true
sort. Ezután minden beírt parancs végre fog hajtódni rákérdezés nélkül.
A telepítéshez a következő sort kell beírni:
conda install pip && pip install pysprint[optional]
(ELAVULT) conda install -c ptrskay pysprint -c conda-forge
Anaconda Promptban a következő paranccsal le is ellenőrizhető, hogy telepítve van-e a csomag.
conda list pysprint
Ez valami hasonlót ad vissza, ha telepítve van:
packages in environment at C:\Users\lp\miniconda3:
Name Version Build Channel
pysprint 0.13.1 py37_0 ptrskay
Ha valamilyen okból nem sikerült, akkor az előbbi parancs egy üres sort fog visszaadni.
A csomag frissítése ugyancsak az Anaconda Prompttal történik (fontos lehet, mivel a jövőben még frissíteni fogom a programot).
A frissíteni a köv. paranccsal lehetséges:
python -m pip install --upgrade pysprint
(ELAVULT) conda update pysprint
A frissítésen és telepítésen kívül az Anaconda Prompt nem szükséges.
1.1.2 Hagyományos Python környezet, PIP¶
Hagyományos Python környezetet használva a telepítés a Parancssorban történik:
python -m pip install pysprint
A frissítéshez tartozó parancs pedig:
python -m pip install --upgrade pysprint
1.2 A development verzió telepítése, contributing¶
Új felhasználók számára a Git verziókövető szoftver ismerete lehet a legnehezebben érthető. Nagyon gyorsan bonyolulttá válhat, de az itt leírtak segíthetnek a folyamat könnyű és gondmentes végrehajtásában. A kód a GitHub-on tárolódik. Ahhoz, hogy a program fejlesztésében részt lehessen venni, szükség van egy GitHub felhasználóra, illetve a Git szoftver telepítésére.
Néhány forrás, ahol részletesebben olvashatunk a Git-ről: * GitHub instructions * NumPy documentation
Az első lépés ezek után egy saját “fork” készítése. A projekt oldalán a jobb felső sarokban a Fork gomb megnyomása után egy másolatot készít a saját felhasználónknak. Ezután a parancssort használva a következőket kell beírnunk:
git clone https://github.com/your-user-name/pysprint.git pysprint-yourname
cd pysprint-yourname
git remote add upstream https://github.com/Ptrskay3/PySprint
Itt természetesen a your-user-name helyére a saját Git felhasználónk nevét kell helyettesíteni. Ez a gépünkre letölti a megfelelő fájlokat a pysprint-yourname könyvtárba és összekapcsolja azt az “upstream” kóddal.
Töltsük le a Rust programozási nyelvet, majd a terminálban váltsunk a nightly verziójára:
rustup toolchain install nightly
rustup override set nightly
Ezután a megfelelő Python környezetet kell beállítanunk. Anaconda esetén ez egyszerű:
A terminált használva először győződjünk meg, hogy az Anaconda legfrissebb verziója van-e telepítve (conda update conda). Ez követően navigáljunk el a pysprint-yourname könyvtárba, ahova a program letöltődött. Itt a parancssorban futtassuk a következőket:
# a környezet létrehozása és aktiválása
conda env create -f environment.yml
conda activate pysprint-dev
# a program telepítése a letöltött fájlokból
python -m pip install -e .[optional]
Ekkor a lokális telepítésből már tudjuk importálni a csomagot:
python # interpreter elindítása
>>> import pysprint as ps
>>> print(ps.__version__)
0.13.1
A Git segítségével különböző ágakkal (branch) lehet dolgozni. Ezek közül a fő ág a master. A master branch-en csakis a production-ready kódot szeretnénk tartani, így ott sose dolgozzunk. Először győződjünk meg, hogy a legfrissebb verzió van nálunk:
git checkout master
git pull upstream master
Készítsünk egy új ágat:
git checkout -b my-new-feature
Ezzel létrehoztuk a my-new-feature ágat és át is váltottunk rá. Ezután elkezdhetjük a kódot írni.
Ha megváltoztattunk bizonyos fájlokat, akkor a git status paranccsal ellenőrizhetjük a fájlok státuszát. Mielőtt a változtatásokat véglegesítenénk előbb mindenképpen futtassuk le az automatizált teszteket, hogy megbizonyosodjuk arról, hogy nem rontottunk el már meglévő funkciókat. Ezt két módon lehetséges a gyökérkönyvtárban:
cd pysprint/tests
pytest -vv
vagy
tox
Amennyiben ezek nem adnak hibát akkor a változtatásokat a saját “fork”-ba fel is tölthetjük a következő módon:
git add -A
git commit -m "some description about what I've done"
git push origin my-new-feature
A manuális környezet beállítása helyett használható a Docker is. A projekt biztosít ehhez a főkönyvtárában egy Dockerfile-t.
1.3 Első lépések¶
Szigorúan véve nem szükséges a numpy és matplotlib importálás ahhoz, hogy a csomag működjön, de a biztonság kedvéért ajánlott előtte azokat is meghívni. Ez után a csomag importálása szokásos módon az
import pysprint as ps
sorral történik. A ps rövidítést én vezettem be, ezt tartottam logikusnak.
[1]:
import numpy as np
import matplotlib.pyplot as plt
import pysprint as ps
A verzió számát a python csomagoknál megszokott
ps.__version__
rövidítéssel érhetjük el. A későbbiekben fontos lesz, mivel néhány kisebb hiba javításra kerülhet.
[2]:
print('verzió: ', ps.__version__)
print('szerző: ', ps.__author__)
verzió: e29766d-dirty
szerző: Leéh Péter
A saját telepítésemnél a verzió száma más logikán alapul, normál telepítésnél rendes verziószámot fog kiírni, pl. 0.13.1.
1.4 Hibák jelentése¶
A programhoz tartozik a print_info függvény, amivel minden fontos információt kiír a környezetről, ami a problémák megoldásánál hasznos lehet. Hibát jelenteni a szoftver GitHub oldalán az issue fül alatt lehetséges, vagy a leeh123peter@gmail.com e-mail címen.
[3]:
ps.print_info()
PYSPRINT ANALYSIS TOOL
SYSTEM
----------------------
python : 3.7.6.final.0
python-bits: 64
OS : Windows
OS-release : 10
Version : 10.0.18362
machine : AMD64
processor : Intel64 Family 6 Model 158 Stepping 9, GenuineIntel
byteorder : little
DEPENDENCY
----------------------
pysprint : e29766d-dirty
numpy : 1.19.4
scipy : 1.5.4
matplotlib : 3.3.3
pandas : 1.1.4
pytest : 6.1.1
lmfit : 1.0.1
numba : 0.50.1
IPython : 7.12.0
jinja2 : 2.11.2
dask : 2.29.0
sklearn : 0.23.2
ADDITIONAL
----------------------
Conda-env : False
IPython : True
Spyder : False
Rust-ext : True
1.5 Mértékegységek¶
Először fontos kikötni a mértékegységeket: hullámhossztartományban nm, frekvenciatartományban PHz egységekben dolgozik a program. Minden kiértékelés előtt frekvenciatartományba kell váltani az adatsort. Az eredmények ezután fs (és hatványai) egységekben lesznek kiszámítva.
2. Szimulációk¶
Ez egy kevésbé használt funkció, a példák során ezt is fogom használni, így illik ezt is bemutatni. A funkció a következőképpen néz ki (az alapértelmezett értékeivel):
ps.Generator(
start, < -- a spektrum kezdőértéke, hullámhossztartomány esetén nm, frekvenciatartomány esetén PHz
stop, < -- a spektrum végértéke, hullámhossztartomány esetén nm, frekvenciatartomány esetén PHz
center, < -- a központi hullámhossz/frekvencia
delay=0, < -- a karok közti időbeli késleltetés fs egységekben
GD=0,
GDD=0,
TOD=0, < -- a diszperziós együtthatók
FOD=0,
QOD=0,
resolution=0.1, < -- az interferogram felbontása nm egységekben (függetlenül milyen tartományban dolgozunk)
delimiter=",", < -- a mentésnél az alapértelmezett elválasztó
pulse_width=10, < -- az impulzus félértékszélességét szabályzó paraméter fs egységben (lent tau_p)
normalize=False, < -- normálja-e az interferogramot (ha igen, akkor a karok spektrumát is visszaadja)
chirp=0, < -- lineáris csörp paraméter (lent a)
)
A spektrális intenzitás alakja:
\(\LARGE{I(\omega) = C\cdot e^{\frac{(\omega-\omega_0)^2\cdot \tau_p^2}{4 ln2\sqrt{1 + a^2}}}}\)
A használatához először inicializálni kell a Generator objektumot, aztán annak függvényében, hogy milyen tartományban szeretnénk generálni az interferogramot két lehetőség van: * ps.Generator.generate_freq() - a frekvenciatartománybeli generálás * ps.Generator.generate_wave() - a hullámhossztartománybeli generálás * ps.Generator.generate() - automatikusan eldönti, hogy milyen tartományban generálja
A következőkben két interferogramot generálok, az egyiket frekvenciatartományban (1 és 4 PHz között 2.355 PHz központi frekvenciával fog egy harmadrendű diszperziót tartalmazó interferogramot generálni 100 fs karok közti késleltetésnél), illetve a másikat hullámhossztartományban (hasonló paraméterekkel, csak 400 és 1000 nm között 800 nm központi hullámhosszal).
[4]:
f = ps.Generator(1, 4, 2.355, 100, TOD=500, pulse_width=5) # <-- itt még nem számolja ki a spektrumot
f.generate_freq() # < -- ezzel a sorral már kiszámolja a spektrumot
g = ps.Generator(400, 1000, 800, 100, TOD=500, normalize=True) # <-- hasonlóan itt még nem számolja ki a spektrumot
g.generate_wave() # < -- ezzel a sorral már kiszámolja a spektrumot
Ezzel létrehoztuk a g és f generátor objektumokat és generáltuk is a hozzájuk tartozó spektrumokat. Néhány funkciója a Generator objektumoknak: * ps.Generator.show() <– mutassa a generált interferogramot * ps.Generator.phase_graph() <– mutassa a generált interferogramot együtt a spektrális fázisfüggvénnyel * ps.Generator.save(name) <– elmenti a spektrumot name névvel egy txt fájlba * ps.Generator.data <– adja vissza a generált adatokat np.ndarray-ként
Példaként néhányat ezek közül meghívok:
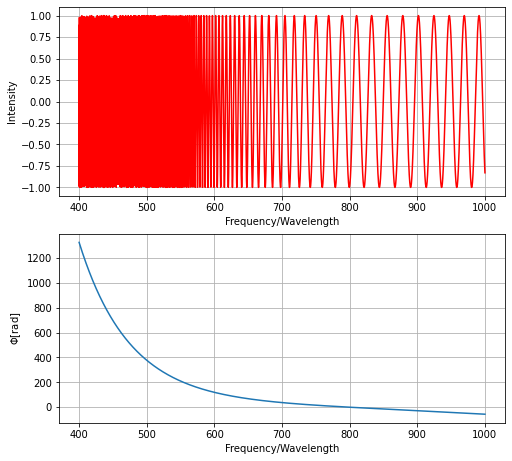
[5]:
g.phase_graph()
[6]:
g.save("ifg")
Successfully saved as ifg.txt.
[7]:
f.show()
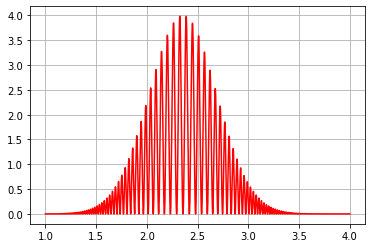
A generált adatok eléréséhez a ps.Generator.data függvényt használhatjuk. Ha normáljuk az interferogramot, akkor ez vissza fogja adni a karok spektrumait is. Minden visszaadott érték np.ndarray típusú.
[8]:
gx, gy, gref, gsam = g.data # a g normált volt, ezért a referencia- és tárgynyaláb spektrumát is visszaadja
[9]:
fx, fy = f.data # az f pedig nem
[10]:
print(type(fy))
<class 'numpy.ndarray'>
3. Alapelemek és koszinusz-függvény illesztéses módszer, CosFitMethod¶
Néhány művelet minden kiértékelési módszernél hasznos lehet, (ilyenek pl. a hullámhossztartományból frekvenciatartományba váltás vagy fordítva, normálás, fontos részek kivágása, stb.) ezért ezt minden objektum eléri. Erről egy külön munkafüzetet készítettem, ahol részletesen leírok mindent. Ez a következő szekcióban érhető el.
A támogatott kiértékelési eljárások:
ps.MinMaxMethod- Minimum-maximum módszerps.CosFitMethod- Fázismodulált koszinusz-függvény illesztéses módszerps.FFTMethod- Fourier-transzformációs módszerps.WFTMethod- Ablakolt Fourier-transzformációs módszer (a 0.12.1 verziótól elérhető)ps.SPPMethod- Állandó fázisú pont módszere
Az alábbi bevezető példában a CosFitMethod-ot fogom használni. Kétféleképpen tölthetjük be az interferogramot: fájlból töltjük be az adatokat, vagy np.ndarray-ban már Pythonban be van töltve nekünk (pl. a szimulációt használtuk) a szükséges spektrum.
[11]:
# 1. mód: np.ndarrayból töltjük be
a = ps.CosFitMethod(gx, gy, gref, gsam) # < -- ezek a fenti szimulációból származnak
# vagy röviden:
# a = ps.CosFitMethod(*g.data)
# 2. mód: fájlból töltjük be
b = ps.CosFitMethod.parse_raw(
'datasets/ifg.trt',
'datasets/ref.trt',
'datasets/sam.trt',
skiprows=8,
meta_len=6,
delimiter=";",
decimal=","
)
c:\pyt\pysprint\pysprint\core\bases\dataset.py:207: PySprintWarning: Extreme values encountered during normalization.
Nan values: 105
Inf values: 1
PySprintWarning
Az parse_raw függvény alapbeállításait a help(ps.Dataset.parse_raw) függvénnyel könnyen átnézhetjük. Például egy tizedespontokat használó, vesszővel elválasztott adatokat tartalmazó fájlt a következőképpen kellene helyesen betölteni:
mm2 = ps.MinMaxMethod.parse_raw('ifg.txt', 'ref.txt', 'sam.txt', decimal='.', sep=',', skiprows=0, meta_len=0)
[12]:
help(ps.CosFitMethod.parse_raw)
Help on method parse_raw in module pysprint.core.bases.dataset:
parse_raw(filename, ref=None, sam=None, skiprows=0, decimal='.', sep=None, delimiter=None, comment=None, usecols=None, names=None, swapaxes=False, na_values=None, skip_blank_lines=True, keep_default_na=False, meta_len=1, errors='raise', callback=None, parent=None, **kwargs) method of pysprint.core.bases._dataset_base._DatasetBase instance
Dataset object alternative constructor.
Helps to load in data just by giving the filenames in
the target directory.
Parameters
----------
filename: `str`
base interferogram
file generated by the spectrometer
ref: `str`, optional
reference arm's spectra
file generated by the spectrometer
sam: `str`, optional
sample arm's spectra
file generated by the spectrometer
skiprows: `int`, optional
Skip rows at the top of the file. Default is `0`.
decimal: `str`, optional
Character recognized as decimal separator in the original dataset.
Often `,` for European data.
Default is `.`.
sep: `str`, optional
The delimiter in the original interferogram file.
Default is `,`.
delimiter: `str`, optional
The delimiter in the original interferogram file.
This is preferred over the `sep` argument if both given.
Default is `,`.
comment: `str`, optional
Indicates remainder of line should not be parsed. If found at the beginning
of a line, the line will be ignored altogether. This parameter must be a
single character. Default is `'#'`.
usecols: list-like or callable, optional
If there a multiple columns in the file, use only a subset of columns.
Default is [0, 1], which will use the first two columns.
names: array-like, optional
List of column names to use. Default is ['x', 'y']. Column marked
with `x` (`y`) will be treated as the x (y) axis. Combined with the
usecols argument it's possible to select data from a large number of
columns.
swapaxes: bool, optional
Whether to swap x and y values in every parsed file. Default is False.
na_values: scalar, str, list-like, or dict, optional
Additional strings to recognize as NA/NaN. If dict passed, specific
per-column NA values. By default the following values are interpreted as
NaN: ‘’, ‘#N/A’, ‘#N/A N/A’, ‘#NA’, ‘-1.#IND’,
‘-1.#QNAN’, ‘-NaN’, ‘-nan’, ‘1.#IND’, ‘1.#QNAN’,
‘<NA>’, ‘N/A’, ‘NA’, ‘NULL’, ‘NaN’, ‘n/a’, ‘nan’, ‘null’.
skip_blank_lines: bool
If True, skip over blank lines rather than interpreting as NaN values.
Default is True.
keep_default_na: bool
Whether or not to include the default NaN values when parsing the data.
Depending on whether na_values is passed in, the behavior changes. Default
is False. More information available at:
https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.read_csv.html
meta_len: `int`, optional
The first `n` lines in the original file containing the meta
information about the dataset. It is parsed to be dict-like.
If the parsing fails, a new entry will be created in the
dictionary with key `unparsed`.
Default is `1`.
errors: string, optional
Determines the way how mismatching sized datacolumns behave.
The default is `raise`, and it will raise on any error.
If set to `force`, it will truncate every array to have the
same shape as the shortest column. It truncates from
the top of the file.
callback : callable, optional
The function that notifies parent objects about SPP related
changes. In most cases the user should leave this empty. The
default callback is only initialized if this object is constructed
by the `pysprint.SPPMethod` object.
parent : any class, optional
The object which handles the callback function. In most cases
the user should leave this empty.
kwargs : dict, optional
The window class to use in WFTMethod. Has no effect while using other
methods. Must be a subclass of pysprint.core.windows.WindowBase.
Minden ilyen objektum különleges print funkcióval rendelkezik. Itt néhány alapvető információt jelenít meg magáról, illetve ha fájlból töltöttük be, akkor kiolvas adatokat egy saját dictionary-be.
[13]:
print(b)
CosFitMethod
----------
Parameters
----------
Datapoints: 2633
Predicted domain: wavelength
Range: from 360.50000 to 1200.25000 nm
Normalized: True
Delay value: Not given
SPP position(s): Not given
----------------------------
Metadata extracted from file
----------------------------
{
"Average": "1 scans",
"Data measured with spectrometer name": "1107006U1",
"Integration time": "2,00 ms",
"Nr of pixels used for smoothing": "0",
"Wave": "Sample",
"comment": "m_ifg 8,740",
"reference_comment": "m_ref 8,740",
"sample_comment": "m_sam 8,740",
"unparsed": [
[
"Timestamp [10 microsec ticks]256373440"
]
]
}
A saját dictionary a meta kulcsszóval érhető el.
[14]:
b.meta
[14]:
{'comment': 'm_ifg 8,740',
'Integration time': '2,00 ms',
'Average': '1 scans',
'Nr of pixels used for smoothing': '0',
'Data measured with spectrometer name': '1107006U1',
'unparsed': [['Timestamp [10 microsec ticks]256373440']],
'Wave': 'Sample',
'reference_comment': 'm_ref 8,740',
'sample_comment': 'm_sam 8,740'}
Akár saját információt is hozzáadhatunk a dictionary adattípusnál megszokott módon:
[15]:
b.meta['fontos_adat_amit_be_szeretnek_allitani'] = 20
[16]:
b.meta
[16]:
{'comment': 'm_ifg 8,740',
'Integration time': '2,00 ms',
'Average': '1 scans',
'Nr of pixels used for smoothing': '0',
'Data measured with spectrometer name': '1107006U1',
'unparsed': [['Timestamp [10 microsec ticks]256373440']],
'Wave': 'Sample',
'reference_comment': 'm_ref 8,740',
'sample_comment': 'm_sam 8,740',
'fontos_adat_amit_be_szeretnek_allitani': 20}
A metaadatok közül talán a comment a legfontosabb, mivel ez az, amit a mérés során mi írunk be. Ezt kiírtani a köv. módon lehet:
[17]:
print(b.meta['comment'])
m_ifg 8,740
Jupyter Notebookban, illetve Jupyter Labon belül az ilyen objektumoknak - a print funkcióhoz hasonló - HTML reprezentációja van:
[18]:
b
[18]:
| CosFitMethod | |
| Parameters | |
| Datapoints | 2633 |
| Predicted domain | wavelength |
| Range min | 360.50000 nm |
| Range max | 1200.25000 nm |
| Normalized | True |
| Delay value | Not given |
| SPP position(s) | Not given |
| Metadata | |
| comment | m_ifg 8,740 |
|---|---|
| Integration time | 2,00 ms |
| Average | 1 scans |
| Nr of pixels used for smoothing | 0 |
| Data measured with spectrometer name | 1107006U1 |
| unparsed | [['Timestamp [10 microsec ticks]256373440']] |
| Wave | Sample |
| reference_comment | m_ref 8,740 |
| sample_comment | m_sam 8,740 |
| fontos_adat_amit_be_szeretnek_allitani | 20 |
Láthattuk a kiíratás során, hogy Predicted domain: wavelength. Frekvenciatartományba váltani a chdomain() függvénnyel lehet. Ekkor újra kiprintelve már a domain frequency-re vált. Megjegyzés: ez csak akkor működik jól, ha PHz és nm egységekben dolgozunk, különben rossz lehet a predikció.
[19]:
b.chdomain()
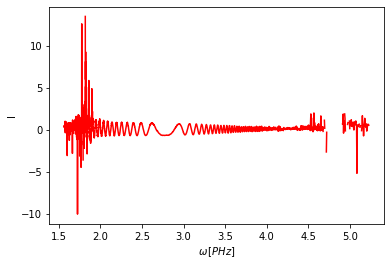
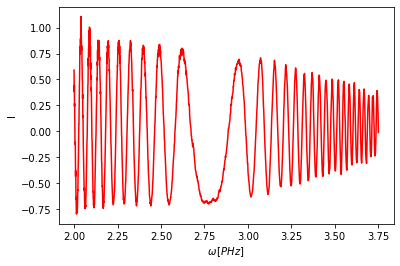
A plot() metódussal meg tudjuk nézni az adatsort. Jupyterben nem szükséges a plt.show() meghívása, de máshol az lehet. Itt látható, hogy a számunkra fontos rész kb. 2.0 és 3.75 PHz között van, a többit nem kívánjuk használni. A kivágás a slice() metódussal lehetséges.
[20]:
b.plot()
[21]:
b.slice(2, 3.75)
b.plot()
Egy ilyen objektumbeli adatokat elérni - a Generatorhoz hasonlóan - a data metódussal lehet. Ez egy pandas.DataFrame objektumot ad vissza, aminek a reprezentációja itt Jupyterben szintén HTML:
[22]:
b.data
[22]:
| x | y | |
|---|---|---|
| 0 | 3.750800 | -0.009973 |
| 1 | 3.748337 | 0.198811 |
| 2 | 3.745877 | 0.322196 |
| 3 | 3.743420 | 0.393736 |
| 4 | 3.740892 | 0.299712 |
| ... | ... | ... |
| 1358 | 2.002649 | 0.240820 |
| 1359 | 2.001989 | 0.446164 |
| 1360 | 2.001330 | 0.380374 |
| 1361 | 2.000671 | 0.550828 |
| 1362 | 1.999991 | 0.589695 |
1363 rows × 2 columns
Ha pl. szeretnénk csak az y értékeket kinyerni np.ndarray formában, akkor azt megtehetjük így:
[23]:
y_ertekei = b.data.y.values
print(type(y_ertekei))
<class 'numpy.ndarray'>
[24]:
print(y_ertekei)
[-0.009973 0.19881073 0.32219592 ... 0.3803739 0.55082753
0.58969469]
A fázismodulált koszinusz-függvény illesztéses módszernél megjelenik néhány specifikus elem. Az első ilyen a GD_lookup(reference_point, engine='cwt', silent=False, **kwargs). Ez a függvény a reference_point környezetében egymást követő minimumok/maximumok távolságát vizsgálja, majd a \(2\pi\)-t osztja ezeknek a távolságoknak az átlagával. Ez jól becsüli a GD-t, amennyiben a szélsőértékeket relatíve pontosan találja meg a függvény. Ennek testreszabására való az engine argumentum,
illetve a további **kwargs keyword argumentumok. Ebben a példában az alapbeállítás megfelelő, így nem módosítok rajta.
[25]:
b.GD_lookup(reference_point=2.355)
The predicted GD is ± 90.17543 fs based on reference point of 2.35500.
Ezt lefuttatva a program már be is állította az illesztésnél a GD kezdőértékét. Természetesen előfordulhat, hogy manuálisan szeretnénk értéket megadni, ezt a
b.guess_GD(szám)
sorral lehetséges. A további együtthatók kezdőértékeit találgatni a GD-vel azonos séma alapján tudjuk:
b.guess_GDD(másik szám)
b.guess_TOD(harmadik szám)
stb.
Koszinusz-függvény illesztésénél két módon végezhetjük el a kiértékelést, a hagyományos illesztési eljárással, vagy a szakdolgozatomban leírt szukcesszív illesztést használva. Először a hagyományost mutatom be. Ekkor relatíve jó kezdeti tippeket kell adnunk a paramétereknek, hogy megfelelően jól közelítse az illesztés az eredeti adatpontokat. Ezt ennél a példánál - ismerve a valódi értékeket - megadom a kezdőértékeket, ezután a set_max_order(order) függvénnyel megadom, hogy maximum
hányadrendű diszperziót keressen. Ezután futtatom a calculate(reference_point) metódust, ami a kiértékelést végzi.
[26]:
b.guess_GD(83) # az előbbi GD = 90.17543 nem elég jó tipp, azzal pontatlan az illesztés
b.guess_GDD(-170)
b.guess_TOD(-115)
b.set_max_order(3)
b.calculate(reference_point=2.355);
Ezután a plot_result() függvényt hívásával az illesztést meg is tekinthetjük.
[27]:
b.plot_result()
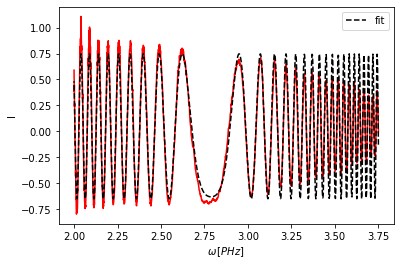
Az általam írt optimalizáló szukcesszív módszert az
optimizer(reference_point,
order=3,
initial_region_ratio=0.1,
extend_by=0.1,
coef_threshold=0.3,
max_tries=5000,
show_endpoint=True
)
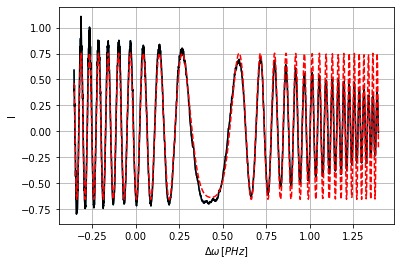
függvény meghívásával érhetjük el. A helyes működéshez az initial_region_ratio argumentum, vagyis a kezdőrégió aránya és az extend_by argumentum, vagyis a tartománynövekedés megfelelő beállítása szükséges. Néhány gyakori hiba és azok megoldása a help(ps.CosFitMethod.optimizer) kiíratásával angol nyelven olvasható. Mivel az előbbi hagyományos kiértékelésnél a kezdőértékeket jól megtippeltem, mielőtt futtatnám a saját optimalizáló algoritmust elrontom a kezdőértékeket, csak a GD-t
hagyom meg. Ebben a példában az alapbeállítás elégséges, így nem változtatok meg semmilyen argumentumot, csak a referencia pontot és a diszperzió rendjét adom meg.
[28]:
b.guess_GDD(1)
b.guess_TOD(1)
b.optimizer(reference_point=2.355, order=3);
Working... \
Kontrasztként bemutatom azt is, hogy mi történik, amikor valamilyen beállítás nem megfelelő. Az extend_by értékét jóval el fogom állítani, vagyis túlságosan gyorsan fog az illesztés régiója nőni, így az eljárás nem fog megfelelő illesztést találni.
[29]:
b.guess_GDD(1)
b.guess_TOD(1)
b.optimizer(reference_point=2.355, order=3, extend_by=0.5);
Working... -
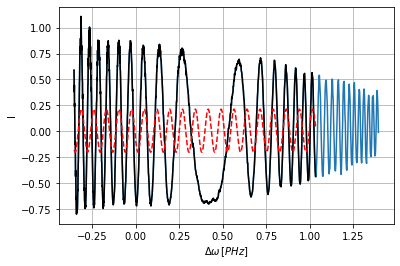
Max tries (currently set to 5000) reached without finding a suitable fit.
c:\program files\python37\lib\site-packages\lmfit\printfuncs.py:179: RuntimeWarning: invalid value encountered in double_scalars
spercent = '({:.2%})'.format(abs(par.stderr/par.value))
Ekkor kékkel az eredeti adatsor, pirossal az illesztett görbe, feketével pedig az aktuális illesztési terület látszódik.